自分のMRI画像をPCで開いて見た
某メーカーのMRIで、自分の頭の撮像をしてもらいました。
コンソールのイメージはだいたいこんな感じ。PCのモニタ上でシーケンスの選択や撮像範囲の選択、画像処理(トリミングなど)を行っていきます。
今回はDiffusion,T1WI,T2WI,3DTOFMRAを撮像してもらいました。撮像されたMRI画像はCD-Rに焼いてもらい、家に持って帰りました。
これがDICOMのメタデータの内容です。モダリティ、撮像シーケンス、TR,TEなど様々なデータが表示されています。
MRI画像のCD-Rのフォルダ構造はこんな感じでした。
医用画像は通常の拡張子ではなく、DICOMという形式なので、専用のビューワーを使わないと不便です。今回、自分はMacのOsirixを用いました。Free版がダウンロード出来ます。
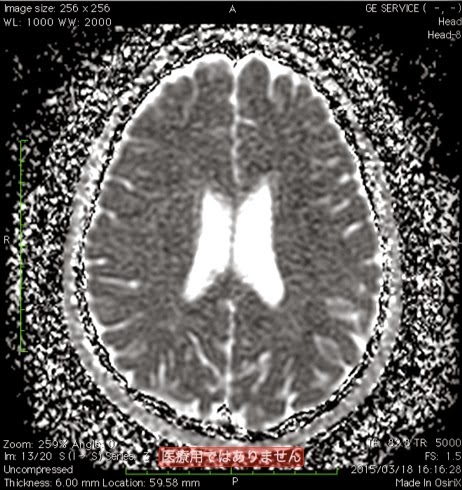
ビューワーで見る自分の脳MRI画像はこんな感じでした。(左がDiffusion,右がMRA)
Diffusionが6mmの40スライス、T1,T2が6mmの20スライスであるのに対して、MRAは1.2mmの164スライスでした。これは、そもそもMRAのシーケンスはGREで、GREのスライスから血流の高信号だけをMIP処理で取り出したのがMRAです。3次元で観察出来るようにする為にも沢山のスライスが必要なんですね。
(因みにT1,T2画像でMPRをやろうとすると、ステアステップが出て何がなんだかよく分かりませんでした。)
OsirixのPluginをダウンロードして、ADCmapを開くことも出来ます。
ADCmapはプロトンの拡散度合いをマトリクス上で表示したものです。DWIでは拡散してないところが高信号になりますが、T2強調の影響を受けているので、ADCmapを確認するとより正確に調べることが出来る様です。
さて、自分の画像を調べたところ、左の側脳室がやや拡大しているようでした。自分の脳に異常が無いか、もう少しきちんと調べたら、紹介しようと思います。
.jpg)







コメント
コメントを投稿